食谱 - 用它做些有趣的事
Biopython 现在有两个“食谱”示例集合 - 本章(已包含在本教程中多年,并且逐渐增长),以及 https://biopython.pythonlang.cn/wiki/Category:Cookbook,它是我们维基上的用户贡献集合。
我们正在努力鼓励 Biopython 用户为维基贡献他们自己的示例。除了帮助社区之外,分享类似示例的一个直接好处是,你还可以从其他 Biopython 用户和开发人员那里获得有关代码的一些反馈 - 这可以帮助你改进所有 Python 代码。
从长远来看,我们可能会最终将本章中的所有示例移至维基,或移至教程中的其他地方。
使用序列文件
本节展示了更多序列输入/输出示例,使用第 序列输入/输出 章中介绍的 Bio.SeqIO 模块。
过滤序列文件
通常你会有一个包含许多序列的大文件(例如,FASTA 文件或基因,或 FASTQ 或 SFF 文件的读取),一个单独的较短的感兴趣序列子集 ID 列表,并且想要为该子集创建一个新的序列文件。
假设 ID 列表位于一个简单的文本文件中,作为每行的第一个词。这可能是一个表格文件,其中第一列是 ID。尝试以下操作
from Bio import SeqIO
input_file = "big_file.sff"
id_file = "short_list.txt"
output_file = "short_list.sff"
with open(id_file) as id_handle:
wanted = set(line.rstrip("\n").split(None, 1)[0] for line in id_handle)
print("Found %i unique identifiers in %s" % (len(wanted), id_file))
records = (r for r in SeqIO.parse(input_file, "sff") if r.id in wanted)
count = SeqIO.write(records, output_file, "sff")
print("Saved %i records from %s to %s" % (count, input_file, output_file))
if count < len(wanted):
print("Warning %i IDs not found in %s" % (len(wanted) - count, input_file))
请注意,我们使用 Python set 而不是 list,这使得测试成员资格更快。
如第 低级 FASTA 和 FASTQ 解析器 节中所述,对于大型 FASTA 或 FASTQ 文件,为了提高速度,最好不要使用高级 SeqIO 接口,而是直接使用字符串。以下示例展示了如何使用 FASTQ 文件进行操作 - 这更复杂
from Bio.SeqIO.QualityIO import FastqGeneralIterator
input_file = "big_file.fastq"
id_file = "short_list.txt"
output_file = "short_list.fastq"
with open(id_file) as id_handle:
# Taking first word on each line as an identifier
wanted = set(line.rstrip("\n").split(None, 1)[0] for line in id_handle)
print("Found %i unique identifiers in %s" % (len(wanted), id_file))
with open(input_file) as in_handle:
with open(output_file, "w") as out_handle:
for title, seq, qual in FastqGeneralIterator(in_handle):
# The ID is the first word in the title line (after the @ sign):
if title.split(None, 1)[0] in wanted:
# This produces a standard 4-line FASTQ entry:
out_handle.write("@%s\n%s\n+\n%s\n" % (title, seq, qual))
count += 1
print("Saved %i records from %s to %s" % (count, input_file, output_file))
if count < len(wanted):
print("Warning %i IDs not found in %s" % (len(wanted) - count, input_file))
生成随机基因组
假设你正在查看基因组序列,寻找一些序列特征 - 也许是极端的局部 GC% 偏差,或可能的限制性酶切位点。一旦你的 Python 代码在真实基因组上运行起来,可能明智的做法是在相同基因组的随机版本上运行相同的搜索,以进行统计分析(毕竟,你发现的任何“特征”都可能仅仅是偶然出现的)。
在本讨论中,我们将使用来自鼠疫耶尔森氏菌微鼠生物变种的 pPCP1 质粒的 GenBank 文件。该文件包含在 Biopython 单元测试中,位于 GenBank 文件夹下,或者你可以从我们的网站获取它,NC_005816.gb。该文件只包含一条记录,因此我们可以使用 Bio.SeqIO.read() 函数将其读取为 SeqRecord。
>>> from Bio import SeqIO
>>> original_rec = SeqIO.read("NC_005816.gb", "genbank")
那么,我们如何生成原始序列的随机洗牌版本呢?我会为此使用内置的 Python random 模块,特别是 random.shuffle 函数 - 但这适用于 Python 列表。我们的序列是一个 Seq 对象,因此为了洗牌它,我们需要将其转换为列表
>>> import random
>>> nuc_list = list(original_rec.seq)
>>> random.shuffle(nuc_list) # acts in situ!
现在,为了使用 Bio.SeqIO 输出洗牌后的序列,我们需要使用这个洗牌后的列表构造一个新的 SeqRecord,它包含一个新的 Seq 对象。为此,我们需要将核苷酸列表(单个字母字符串)转换为一个长字符串 - 使用字符串对象的 join 方法是标准的 Python 方法。
>>> from Bio.Seq import Seq
>>> from Bio.SeqRecord import SeqRecord
>>> shuffled_rec = SeqRecord(
... Seq("".join(nuc_list)), id="Shuffled", description="Based on %s" % original_rec.id
... )
让我们把所有这些部分放在一起,编写一个完整的 Python 脚本,该脚本生成一个包含原始序列的 30 个随机洗牌版本的 FASTA 文件。
这个第一个版本只使用一个大的 for 循环,并逐个写入记录(使用第 将你的 SeqRecord 对象作为格式化字符串获取 节中介绍的 SeqRecord 的 format 方法)
import random
from Bio.Seq import Seq
from Bio.SeqRecord import SeqRecord
from Bio import SeqIO
original_rec = SeqIO.read("NC_005816.gb", "genbank")
with open("shuffled.fasta", "w") as output_handle:
for i in range(30):
nuc_list = list(original_rec.seq)
random.shuffle(nuc_list)
shuffled_rec = SeqRecord(
Seq("".join(nuc_list)),
id="Shuffled%i" % (i + 1),
description="Based on %s" % original_rec.id,
)
output_handle.write(shuffled_rec.format("fasta"))
我个人更喜欢以下版本,它使用一个函数来洗牌记录,并使用生成器表达式而不是 for 循环
import random
from Bio.Seq import Seq
from Bio.SeqRecord import SeqRecord
from Bio import SeqIO
def make_shuffle_record(record, new_id):
nuc_list = list(record.seq)
random.shuffle(nuc_list)
return SeqRecord(
Seq("".join(nuc_list)),
id=new_id,
description="Based on %s" % original_rec.id,
)
original_rec = SeqIO.read("NC_005816.gb", "genbank")
shuffled_recs = (
make_shuffle_record(original_rec, "Shuffled%i" % (i + 1)) for i in range(30)
)
SeqIO.write(shuffled_recs, "shuffled.fasta", "fasta")
翻译 FASTA 文件中的 CDS 条目
假设你有一个包含某个生物体的 CDS 条目的输入文件,并且你想要生成一个包含其蛋白质序列的新 FASTA 文件。即,获取原始文件中的每个核苷酸序列,并对其进行翻译。回到第 翻译 节,我们看到了如何使用 Seq 对象的 translate method,以及可选的 cds 参数,该参数可以正确翻译备选起始密码子。
我们可以将它与 Bio.SeqIO 相结合,如第 将一系列序列转换为其反向互补序列 节中的反向互补示例所示。关键点是,对于每个核苷酸 SeqRecord,我们需要创建一个蛋白质 SeqRecord - 并注意命名它。
你可以编写自己的函数来完成此操作,为你的序列选择合适的蛋白质标识符,以及合适的遗传密码。在本例中,我们只使用默认表,并在标识符前添加前缀
from Bio.SeqRecord import SeqRecord
def make_protein_record(nuc_record):
"""Returns a new SeqRecord with the translated sequence (default table)."""
return SeqRecord(
seq=nuc_record.seq.translate(cds=True),
id="trans_" + nuc_record.id,
description="translation of CDS, using default table",
)
然后我们可以使用此函数将输入的核苷酸记录转换为蛋白质记录,准备输出。一种优雅且内存高效的方法是使用生成器表达式
from Bio import SeqIO
proteins = (
make_protein_record(nuc_rec)
for nuc_rec in SeqIO.parse("coding_sequences.fasta", "fasta")
)
SeqIO.write(proteins, "translations.fasta", "fasta")
这应该适用于任何包含完整编码序列的 FASTA 文件。如果你正在处理部分编码序列,你可能更愿意在上面的示例中使用 nuc_record.seq.translate(to_stop=True),因为这不会检查有效的起始密码子等。
将 FASTA 文件中的序列转换为大写
通常你会从合作者那里获得 FASTA 文件格式的数据,有时序列可能是大小写混合的。在某些情况下,这是故意的(例如,低质量区域为小写),但通常并不重要。你可能想要编辑该文件以使所有内容保持一致(例如,全部大写),你可以使用 SeqRecord 对象的 upper() 方法(在 Biopython 1.55 中添加)轻松完成此操作
from Bio import SeqIO
records = (rec.upper() for rec in SeqIO.parse("mixed.fas", "fasta"))
count = SeqIO.write(records, "upper.fas", "fasta")
print("Converted %i records to upper case" % count)
这是如何工作的?第一行只是导入 Bio.SeqIO 模块。第二行是最有趣的 - 这是一个 Python 生成器表达式,它提供从输入文件 (mixed.fas) 解析的每个记录的大写版本。在第三行中,我们将此生成器表达式提供给 Bio.SeqIO.write() 函数,它将新的大写记录保存到我们的输出文件 (upper.fas) 中。
我们使用生成器表达式(而不是列表或列表推导)的原因是,这意味着一次只保留一条记录在内存中。如果你正在处理包含数百万条记录的大文件,这可能非常重要。
排序序列文件
假设你想要按长度对序列文件进行排序(例如,来自组装的一组拼接体),并且你正在使用 Bio.SeqIO 可以读取、写入(和索引)的 FASTA 或 FASTQ 等文件格式。
如果文件足够小,你可以一次将所有文件加载到内存中,作为一个 SeqRecord 对象列表,对列表进行排序,然后保存它
from Bio import SeqIO
records = list(SeqIO.parse("ls_orchid.fasta", "fasta"))
records.sort(key=lambda r: len(r))
SeqIO.write(records, "sorted_orchids.fasta", "fasta")
唯一巧妙的部分是指定如何排序记录的比较方法(这里我们按长度对它们进行排序)。如果你想要最长的记录排在最前面,你可以翻转比较或使用 reverse 参数
from Bio import SeqIO
records = list(SeqIO.parse("ls_orchid.fasta", "fasta"))
records.sort(key=lambda r: -len(r))
SeqIO.write(records, "sorted_orchids.fasta", "fasta")
这非常直观——但如果你的文件非常大,无法像这样全部加载到内存中会怎么样?例如,你可能有一些需要按长度排序的下一代测序读段。这可以使用 Bio.SeqIO.index() 函数来解决。
from Bio import SeqIO
# Get the lengths and ids, and sort on length
len_and_ids = sorted(
(len(rec), rec.id) for rec in SeqIO.parse("ls_orchid.fasta", "fasta")
)
ids = reversed([id for (length, id) in len_and_ids])
del len_and_ids # free this memory
record_index = SeqIO.index("ls_orchid.fasta", "fasta")
records = (record_index[id] for id in ids)
SeqIO.write(records, "sorted.fasta", "fasta")
首先,我们使用 Bio.SeqIO.parse() 扫描文件一次,将记录标识符及其长度记录在一个元组列表中。然后,我们对这个列表进行排序,使其按长度排序,并丢弃长度。使用这个排序后的标识符列表,Bio.SeqIO.index() 允许我们逐个检索记录,并将它们传递给 Bio.SeqIO.write() 进行输出。
这些例子都使用 Bio.SeqIO 将记录解析为 SeqRecord 对象,然后使用 Bio.SeqIO.write() 输出。如果你想排序 Bio.SeqIO.write() 不支持的文件格式,比如纯文本 SwissProt 格式,该怎么办?这里有一个使用 get_raw() 方法的替代解决方案,该方法在 Biopython 1.54 中添加到 Bio.SeqIO.index()(参见第 获取记录的原始数据 节)。
from Bio import SeqIO
# Get the lengths and ids, and sort on length
len_and_ids = sorted(
(len(rec), rec.id) for rec in SeqIO.parse("ls_orchid.fasta", "fasta")
)
ids = reversed([id for (length, id) in len_and_ids])
del len_and_ids # free this memory
record_index = SeqIO.index("ls_orchid.fasta", "fasta")
with open("sorted.fasta", "wb") as out_handle:
for id in ids:
out_handle.write(record_index.get_raw(id))
请注意,从 Python 3 开始,我们必须以二进制模式打开文件以进行写入,因为 get_raw() 方法返回 bytes 对象。
作为一个额外的好处,因为它不会将数据解析为 SeqRecord 对象第二次,所以应该更快。如果你只想将它用于 FASTA 格式,我们可以通过使用低级 FASTA 解析器获取记录标识符和长度来进一步加快速度。
from Bio.SeqIO.FastaIO import SimpleFastaParser
from Bio import SeqIO
# Get the lengths and ids, and sort on length
with open("ls_orchid.fasta") as in_handle:
len_and_ids = sorted(
(len(seq), title.split(None, 1)[0])
for title, seq in SimpleFastaParser(in_handle)
)
ids = reversed([id for (length, id) in len_and_ids])
del len_and_ids # free this memory
record_index = SeqIO.index("ls_orchid.fasta", "fasta")
with open("sorted.fasta", "wb") as out_handle:
for id in ids:
out_handle.write(record_index.get_raw(id))
针对 FASTQ 文件的简单质量过滤
FASTQ 文件格式起源于桑格研究院,现在被广泛用于存储核苷酸测序读段及其质量评分。FASTQ 文件(以及相关的 QUAL 文件)是逐字母注释的一个很好的例子,因为序列中的每个核苷酸都有一个相关的质量评分。任何逐字母注释都保存在 SeqRecord 对象的 letter_annotations 字典中,作为列表、元组或字符串(具有与序列长度相同的元素数量)。
一个常见的任务是获取大量测序读段,并根据其质量评分对其进行过滤(或裁剪)。以下示例非常简单,但应该说明在 SeqRecord 对象中处理质量数据的基本原理。我们在这里要做的是读取一个 FASTQ 数据文件,并对其进行过滤,以选择 PHRED 质量评分均高于某个阈值(这里为 20)的记录。
对于这个例子,我们将使用从 ENA 序列读取档案下载的一些真实数据,ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR020/SRR020192/SRR020192.fastq.gz(2MB),解压缩后得到一个 19MB 的文件 SRR020192.fastq。这是从感染病毒的加州海狮身上获得的 Roche 454 GS FLX 单端数据(有关详细信息,请参见 https://www.ebi.ac.uk/ena/data/view/SRS004476)。
首先,让我们统计读段数。
from Bio import SeqIO
count = 0
for rec in SeqIO.parse("SRR020192.fastq", "fastq"):
count += 1
print("%i reads" % count)
现在让我们进行一个简单的过滤,以确保最小 PHRED 质量为 20。
from Bio import SeqIO
good_reads = (
rec
for rec in SeqIO.parse("SRR020192.fastq", "fastq")
if min(rec.letter_annotations["phred_quality"]) >= 20
)
count = SeqIO.write(good_reads, "good_quality.fastq", "fastq")
print("Saved %i reads" % count)
这从 \(41892\) 个读段中只提取了 \(14580\) 个读段。更合理的做法是将读段进行质量修剪,但这只是一个示例。
FASTQ 文件可能包含数百万个条目,因此最好避免将它们全部一次性加载到内存中。此示例使用生成器表达式,这意味着一次只创建一个 SeqRecord 对象,避免任何内存限制。
请注意,使用低级 FastqGeneralIterator 解析器在这里会更快(参见第 低级 FASTA 和 FASTQ 解析器 节),但它不会将质量字符串转换为整数评分。
修剪引物序列
对于这个例子,我们将假装 GATGACGGTGT 是我们想要在一些 FASTQ 格式的读段数据中查找的 5’ 引物序列。和上面的例子一样,我们将使用从 ENA 下载的 SRR020192.fastq 文件 (ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR020/SRR020192/SRR020192.fastq.gz)。
通过使用主要的 Bio.SeqIO 接口,相同的方法适用于任何其他受支持的文件格式(例如 FASTA 文件)。但是,对于大型 FASTQ 文件,使用低级 FastqGeneralIterator 解析器在这里会更快(参见前面的示例以及第 低级 FASTA 和 FASTQ 解析器 节)。
这段代码使用 Bio.SeqIO 和生成器表达式(以避免将所有序列一次性加载到内存中),以及 Seq 对象的 startswith 方法来判断读段是否以引物序列开头。
from Bio import SeqIO
primer_reads = (
rec
for rec in SeqIO.parse("SRR020192.fastq", "fastq")
if rec.seq.startswith("GATGACGGTGT")
)
count = SeqIO.write(primer_reads, "with_primer.fastq", "fastq")
print("Saved %i reads" % count)
这应该从 SRR014849.fastq 中找到 \(13819\) 个读段,并将它们保存到一个新的 FASTQ 文件 with_primer.fastq 中。
现在假设你想要创建一个包含这些读段的 FASTQ 文件,但要删除引物序列?这只是一个小的改动,因为我们可以对 SeqRecord 进行切片(参见第 对 SeqRecord 进行切片 节)以删除前十一个字母(我们引物的长度)。
from Bio import SeqIO
trimmed_primer_reads = (
rec[11:]
for rec in SeqIO.parse("SRR020192.fastq", "fastq")
if rec.seq.startswith("GATGACGGTGT")
)
count = SeqIO.write(trimmed_primer_reads, "with_primer_trimmed.fastq", "fastq")
print("Saved %i reads" % count)
同样,这应该从 SRR020192.fastq 中提取 \(13819\) 个读段,但这次要删除前十个字符,并将它们保存到另一个新的 FASTQ 文件 with_primer_trimmed.fastq 中。
现在,假设你想要创建一个新的 FASTQ 文件,其中这些读段的引物被删除,但所有其他读段都保留原样?如果我们仍然想要使用生成器表达式,定义我们自己的修剪函数可能是最清晰的。
from Bio import SeqIO
def trim_primer(record, primer):
if record.seq.startswith(primer):
return record[len(primer) :]
else:
return record
trimmed_reads = (
trim_primer(record, "GATGACGGTGT")
for record in SeqIO.parse("SRR020192.fastq", "fastq")
)
count = SeqIO.write(trimmed_reads, "trimmed.fastq", "fastq")
print("Saved %i reads" % count)
这需要更长的时间,因为这次输出文件包含所有 \(41892\) 个读段。同样,我们使用了生成器表达式来避免任何内存问题。你也可以使用生成器函数而不是生成器表达式。
from Bio import SeqIO
def trim_primers(records, primer):
"""Removes perfect primer sequences at start of reads.
This is a generator function, the records argument should
be a list or iterator returning SeqRecord objects.
"""
len_primer = len(primer) # cache this for later
for record in records:
if record.seq.startswith(primer):
yield record[len_primer:]
else:
yield record
original_reads = SeqIO.parse("SRR020192.fastq", "fastq")
trimmed_reads = trim_primers(original_reads, "GATGACGGTGT")
count = SeqIO.write(trimmed_reads, "trimmed.fastq", "fastq")
print("Saved %i reads" % count)
如果想要做一些更复杂的事情,例如只保留一些记录,这种形式会更灵活,如下一示例所示。
修剪接头序列
这本质上是对前一个示例的简单扩展。我们将假装 GATGACGGTGT 是某些 FASTQ 格式读段数据中的接头序列,再次是来自 NCBI 的 SRR020192.fastq 文件 (ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR020/SRR020192/SRR020192.fastq.gz)。
然而,这次我们将寻找任何位置的序列,而不仅仅是在开头。
from Bio import SeqIO
def trim_adaptors(records, adaptor):
"""Trims perfect adaptor sequences.
This is a generator function, the records argument should
be a list or iterator returning SeqRecord objects.
"""
len_adaptor = len(adaptor) # cache this for later
for record in records:
index = record.seq.find(adaptor)
if index == -1:
# adaptor not found, so won't trim
yield record
else:
# trim off the adaptor
yield record[index + len_adaptor :]
original_reads = SeqIO.parse("SRR020192.fastq", "fastq")
trimmed_reads = trim_adaptors(original_reads, "GATGACGGTGT")
count = SeqIO.write(trimmed_reads, "trimmed.fastq", "fastq")
print("Saved %i reads" % count)
因为我们在本示例中使用的是 FASTQ 输入文件,所以 SeqRecord 对象具有质量评分的逐字母注释。通过对 SeqRecord 对象进行切片,将使用相应的评分对修剪后的记录进行评分,因此我们可以将它们输出为 FASTQ 文件。
与前面只在每个读段开头查找引物/接头的示例的输出相比,你可能会发现一些修剪后的读段在修剪后非常短(例如,如果接头是在中间而不是靠近开头找到的)。所以,让我们也添加一个最小长度要求。
from Bio import SeqIO
def trim_adaptors(records, adaptor, min_len):
"""Trims perfect adaptor sequences, checks read length.
This is a generator function, the records argument should
be a list or iterator returning SeqRecord objects.
"""
len_adaptor = len(adaptor) # cache this for later
for record in records:
len_record = len(record) # cache this for later
if len(record) < min_len:
# Too short to keep
continue
index = record.seq.find(adaptor)
if index == -1:
# adaptor not found, so won't trim
yield record
elif len_record - index - len_adaptor >= min_len:
# after trimming this will still be long enough
yield record[index + len_adaptor :]
original_reads = SeqIO.parse("SRR020192.fastq", "fastq")
trimmed_reads = trim_adaptors(original_reads, "GATGACGGTGT", 100)
count = SeqIO.write(trimmed_reads, "trimmed.fastq", "fastq")
print("Saved %i reads" % count)
通过更改格式名称,你可以将其应用于 FASTA 文件。这段代码还可以扩展为进行模糊匹配而不是精确匹配(也许使用成对比对,或者考虑读段质量评分),但这会慢得多。
转换 FASTQ 文件
回到第 在序列文件格式之间转换 节,我们展示了如何使用 Bio.SeqIO 在两种文件格式之间进行转换。在这里,我们将深入探讨与第二代 DNA 测序中使用的 FASTQ 文件相关的更多细节。请参考 Cock 等人 (2010) [Cock2010] 以获得更详细的描述。FASTQ 文件存储 DNA 序列(作为字符串)和相关的读段质量。
PHRED 评分(在大多数 FASTQ 文件中使用,以及在 QUAL 文件、ACE 文件和 SFF 文件中使用)已成为表示给定碱基测序错误概率(这里用 \(P_e\) 表示)的实际标准,使用简单的以十为底的对数变换。
这意味着一个错误读段 (\(P_e = 1\)) 的 PHRED 质量为 \(0\),而一个非常好的读段,如 \(P_e = 0.00001\) 的 PHRED 质量为 \(50\)。虽然对于原始测序数据,高于此的质量很少见,但在读段比对或组装等后处理中,高达约 \(90\) 的质量是可能的(事实上,MAQ 工具允许 PHRED 评分在 0 到 93(含)的范围内)。
FASTQ 格式有可能成为存储测序读段的字母和质量评分的实际标准,并将它们存储在一个单独的纯文本文件中。唯一的障碍是,FASTQ 格式至少有三个版本,它们不兼容且难以区分……
最初的桑格研究院 FASTQ 格式使用 ASCII 偏移量为 33 的 PHRED 质量编码。NCBI 在其短读段档案中使用这种格式。我们在
Bio.SeqIO中将其称为fastq(或fastq-sanger)格式。Solexa(后来被 Illumina 收购)推出了自己的版本,使用 ASCII 偏移量为 64 的 Solexa 质量编码。我们将其称为
fastq-solexa格式。Illumina 管道 1.3 及更高版本生成使用 PHRED 质量编码的 FASTQ 文件(这更加一致),但使用 ASCII 偏移量为 64 编码。我们将其称为
fastq-illumina格式。
Solexa 质量分数使用不同的对数变换定义
鉴于 Solexa/Illumina 现在已在其 1.3 版本的管道中使用 PHRED 分数,Solexa 质量分数将逐渐不再使用。如果将错误估计(\(P_e\))等同起来,这两个方程允许在两个评分系统之间进行转换 - Biopython 在 Bio.SeqIO.QualityIO 模块中包含了执行此操作的函数,如果您使用 Bio.SeqIO 将旧的 Solexa/Illumina 文件转换为标准 Sanger FASTQ 文件,则会调用这些函数
from Bio import SeqIO
SeqIO.convert("solexa.fastq", "fastq-solexa", "standard.fastq", "fastq")
如果要转换新的 Illumina 1.3+ FASTQ 文件,唯一改变的是 ASCII 偏移量,因为尽管编码不同,但分数都是 PHRED 质量
from Bio import SeqIO
SeqIO.convert("illumina.fastq", "fastq-illumina", "standard.fastq", "fastq")
注意,像这样使用 Bio.SeqIO.convert() 比组合使用 Bio.SeqIO.parse() 和 Bio.SeqIO.write() 快得多,因为优化后的代码用于在 FASTQ 变体(以及 FASTQ 到 FASTA 转换)之间进行转换。
对于高质量的读取,PHRED 和 Solexa 分数大致相等,这意味着由于 fasta-solexa 和 fastq-illumina 格式都使用 64 的 ASCII 偏移量,因此文件几乎相同。这是 Illumina 故意做出的设计选择,这意味着期望旧 fasta-solexa 风格文件的应用程序可能可以使用新的 fastq-illumina 文件(对于好的数据)。当然,这两个变体与 Sanger、NCBI 及其他地方使用的原始 FASTQ 标准(格式名称 fastq 或 fastq-sanger)有很大不同。
有关更多详细信息,请参阅内置帮助(也在 Bio.SeqIO.QualityIO 中)
>>> from Bio.SeqIO import QualityIO
>>> help(QualityIO)
将 FASTA 和 QUAL 文件转换为 FASTQ 文件
FASTQ 文件同时包含序列及其质量字符串。FASTA 文件只包含序列,而 QUAL 文件只包含质量。因此,单个 FASTQ 文件可以转换为或从成对的 FASTA 和 QUAL 文件进行转换。
从 FASTQ 到 FASTA 很容易
from Bio import SeqIO
SeqIO.convert("example.fastq", "fastq", "example.fasta", "fasta")
从 FASTQ 到 QUAL 也很容易
from Bio import SeqIO
SeqIO.convert("example.fastq", "fastq", "example.qual", "qual")
但是,反过来有点棘手。可以使用 Bio.SeqIO.parse() 迭代单个文件中的记录,但在这种情况下,我们有两个输入文件。有几种策略可以实现,但假设这两个文件确实是成对的,最节省内存的方法是同时遍历两个文件。代码有点复杂,因此我们在 Bio.SeqIO.QualityIO 模块中提供了一个名为 PairedFastaQualIterator 的函数来执行此操作。这需要两个句柄(FASTA 文件和 QUAL 文件)并返回 SeqRecord 迭代器
from Bio.SeqIO.QualityIO import PairedFastaQualIterator
for record in PairedFastaQualIterator(open("example.fasta"), open("example.qual")):
print(record)
此函数将检查 FASTA 和 QUAL 文件是否一致(例如,记录顺序相同,并且序列长度相同)。可以将它与 Bio.SeqIO.write() 函数结合使用,将一对 FASTA 和 QUAL 文件转换为单个 FASTQ 文件
from Bio import SeqIO
from Bio.SeqIO.QualityIO import PairedFastaQualIterator
with open("example.fasta") as f_handle, open("example.qual") as q_handle:
records = PairedFastaQualIterator(f_handle, q_handle)
count = SeqIO.write(records, "temp.fastq", "fastq")
print("Converted %i records" % count)
索引 FASTQ 文件
FASTQ 文件通常非常大,包含数百万个读取。由于数据量巨大,无法一次将所有记录加载到内存中。这就是为什么上面的示例(过滤和修剪)遍历文件,一次只查看一个 SeqRecord。
但是,有时不能使用大循环或迭代器 - 可能需要随机访问读取。这里 Bio.SeqIO.index() 函数可能会非常有用,因为它允许您通过名称访问 FASTQ 文件中的任何读取(请参阅第 Sequence files as Dictionaries – Indexed files 节)。
我们再次使用 ENA 中的 SRR020192.fastq 文件 (ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR020/SRR020192/SRR020192.fastq.gz),虽然这实际上是一个相当小的 FASTQ 文件,包含不到 \(50,000\) 个读取
>>> from Bio import SeqIO
>>> fq_dict = SeqIO.index("SRR020192.fastq", "fastq")
>>> len(fq_dict)
41892
>>> list(fq_dict.keys())[:4]
['SRR020192.38240', 'SRR020192.23181', 'SRR020192.40568', 'SRR020192.23186']
>>> fq_dict["SRR020192.23186"].seq
Seq('GTCCCAGTATTCGGATTTGTCTGCCAAAACAATGAAATTGACACAGTTTACAAC...CCG')
在对包含七百万个读取的 FASTQ 文件进行测试时,索引大约花费了一分钟,但记录访问几乎是即时的。
姊妹函数 Bio.SeqIO.index_db() 允许您将索引保存到 SQLite3 数据库文件中以供近乎即时的重复使用 - 请参阅第 Sequence files as Dictionaries – Indexed files 节以获取更多详细信息。
第 Sorting a sequence file 节中的示例展示了如何使用 Bio.SeqIO.index() 函数对大型 FASTA 文件进行排序 - 这也可以用于 FASTQ 文件。
转换 SFF 文件
如果您使用 454(Roche)序列数据,您可能可以访问原始数据作为标准流图格式 (SFF) 文件。这包含序列读取(称为碱基)及其质量分数以及原始流信息。
一个常见的任务是从 SFF 转换为一对 FASTA 和 QUAL 文件,或转换为单个 FASTQ 文件。使用 Bio.SeqIO.convert() 函数(请参阅第 Converting between sequence file formats 节)可以轻松完成这些操作
>>> from Bio import SeqIO
>>> SeqIO.convert("E3MFGYR02_random_10_reads.sff", "sff", "reads.fasta", "fasta")
10
>>> SeqIO.convert("E3MFGYR02_random_10_reads.sff", "sff", "reads.qual", "qual")
10
>>> SeqIO.convert("E3MFGYR02_random_10_reads.sff", "sff", "reads.fastq", "fastq")
10
请记住,convert 函数返回记录数量,在本例中只有十个。这将为您提供未修剪的读取,其中前导和尾随的低质量序列或接头将以小写形式显示。如果需要修剪过的读取(使用 SFF 文件中记录的剪切信息),请使用以下方法
>>> from Bio import SeqIO
>>> SeqIO.convert("E3MFGYR02_random_10_reads.sff", "sff-trim", "trimmed.fasta", "fasta")
10
>>> SeqIO.convert("E3MFGYR02_random_10_reads.sff", "sff-trim", "trimmed.qual", "qual")
10
>>> SeqIO.convert("E3MFGYR02_random_10_reads.sff", "sff-trim", "trimmed.fastq", "fastq")
10
如果您运行 Linux,可以向 Roche 索要其“仪器外”工具的副本(通常称为 Newbler 工具)。这提供了在命令行中执行 SFF 到 FASTA 或 QUAL 转换的另一种方法(但目前不支持 FASTQ 输出),例如:
$ sffinfo -seq -notrim E3MFGYR02_random_10_reads.sff > reads.fasta
$ sffinfo -qual -notrim E3MFGYR02_random_10_reads.sff > reads.qual
$ sffinfo -seq -trim E3MFGYR02_random_10_reads.sff > trimmed.fasta
$ sffinfo -qual -trim E3MFGYR02_random_10_reads.sff > trimmed.qual
Biopython 使用混合大小写序列字符串来表示修剪点的方式,故意模仿了 Roche 工具的行为。
有关 Biopython SFF 支持的更多信息,请参阅内置帮助
>>> from Bio.SeqIO import SffIO
>>> help(SffIO)
识别开放阅读框
识别可能基因的一个非常简单的第一步是寻找开放阅读框 (ORF)。通过此操作,我们在所有六个框中寻找没有终止密码子的长区域 - ORF 只是一个没有框内终止密码子的核苷酸区域。
当然,要找到基因,还需要担心找到起始密码子、可能的启动子 - 在真核生物中,还有内含子需要担心。但是,这种方法在病毒和原核生物中仍然有用。
为了展示如何使用 Biopython 来解决这个问题,我们需要一个要搜索的序列,作为示例,我们将再次使用细菌质粒 - 不过这次我们将从一个没有预先标记基因的普通 FASTA 文件开始:NC_005816.fna。这是一个细菌序列,因此我们将使用 NCBI 密码子表 11(有关翻译,请参阅第 Translation 节)。
>>> from Bio import SeqIO
>>> record = SeqIO.read("NC_005816.fna", "fasta")
>>> table = 11
>>> min_pro_len = 100
这是一个使用 Seq 对象的 split 方法获取六个阅读框中所有可能的 ORF 翻译列表的巧妙技巧
>>> for strand, nuc in [(+1, record.seq), (-1, record.seq.reverse_complement())]:
... for frame in range(3):
... length = 3 * ((len(record) - frame) // 3) # Multiple of three
... for pro in nuc[frame : frame + length].translate(table).split("*"):
... if len(pro) >= min_pro_len:
... print(
... "%s...%s - length %i, strand %i, frame %i"
... % (pro[:30], pro[-3:], len(pro), strand, frame)
... )
...
GCLMKKSSIVATIITILSGSANAASSQLIP...YRF - length 315, strand 1, frame 0
KSGELRQTPPASSTLHLRLILQRSGVMMEL...NPE - length 285, strand 1, frame 1
GLNCSFFSICNWKFIDYINRLFQIIYLCKN...YYH - length 176, strand 1, frame 1
VKKILYIKALFLCTVIKLRRFIFSVNNMKF...DLP - length 165, strand 1, frame 1
NQIQGVICSPDSGEFMVTFETVMEIKILHK...GVA - length 355, strand 1, frame 2
RRKEHVSKKRRPQKRPRRRRFFHRLRPPDE...PTR - length 128, strand 1, frame 2
TGKQNSCQMSAIWQLRQNTATKTRQNRARI...AIK - length 100, strand 1, frame 2
QGSGYAFPHASILSGIAMSHFYFLVLHAVK...CSD - length 114, strand -1, frame 0
IYSTSEHTGEQVMRTLDEVIASRSPESQTR...FHV - length 111, strand -1, frame 0
WGKLQVIGLSMWMVLFSQRFDDWLNEQEDA...ESK - length 125, strand -1, frame 1
RGIFMSDTMVVNGSGGVPAFLFSGSTLSSY...LLK - length 361, strand -1, frame 1
WDVKTVTGVLHHPFHLTFSLCPEGATQSGR...VKR - length 111, strand -1, frame 1
LSHTVTDFTDQMAQVGLCQCVNVFLDEVTG...KAA - length 107, strand -1, frame 2
RALTGLSAPGIRSQTSCDRLRELRYVPVSL...PLQ - length 119, strand -1, frame 2
注意,这里我们从每条链的 5' 端(起始端)开始计算框。有时从正向链的 5' 端(起始端)开始计算更方便。
可以轻松地编辑上面的基于循环的代码,以构建候选蛋白质列表,或将其转换为列表推导。现在,这段代码没有做的一件事是跟踪蛋白质的位置。
可以用多种方法解决这个问题。例如,以下代码跟踪蛋白质计数方面的位置,并通过乘以 3 然后根据框和链进行调整将其转换回父序列
from Bio import SeqIO
record = SeqIO.read("NC_005816.gb", "genbank")
table = 11
min_pro_len = 100
def find_orfs_with_trans(seq, trans_table, min_protein_length):
answer = []
seq_len = len(seq)
for strand, nuc in [(+1, seq), (-1, seq.reverse_complement())]:
for frame in range(3):
trans = nuc[frame:].translate(trans_table)
trans_len = len(trans)
aa_start = 0
aa_end = 0
while aa_start < trans_len:
aa_end = trans.find("*", aa_start)
if aa_end == -1:
aa_end = trans_len
if aa_end - aa_start >= min_protein_length:
if strand == 1:
start = frame + aa_start * 3
end = min(seq_len, frame + aa_end * 3 + 3)
else:
start = seq_len - frame - aa_end * 3 - 3
end = seq_len - frame - aa_start * 3
answer.append((start, end, strand, trans[aa_start:aa_end]))
aa_start = aa_end + 1
answer.sort()
return answer
orf_list = find_orfs_with_trans(record.seq, table, min_pro_len)
for start, end, strand, pro in orf_list:
print(
"%s...%s - length %i, strand %i, %i:%i"
% (pro[:30], pro[-3:], len(pro), strand, start, end)
)
以及输出
NQIQGVICSPDSGEFMVTFETVMEIKILHK...GVA - length 355, strand 1, 41:1109
WDVKTVTGVLHHPFHLTFSLCPEGATQSGR...VKR - length 111, strand -1, 491:827
KSGELRQTPPASSTLHLRLILQRSGVMMEL...NPE - length 285, strand 1, 1030:1888
RALTGLSAPGIRSQTSCDRLRELRYVPVSL...PLQ - length 119, strand -1, 2830:3190
RRKEHVSKKRRPQKRPRRRRFFHRLRPPDE...PTR - length 128, strand 1, 3470:3857
GLNCSFFSICNWKFIDYINRLFQIIYLCKN...YYH - length 176, strand 1, 4249:4780
RGIFMSDTMVVNGSGGVPAFLFSGSTLSSY...LLK - length 361, strand -1, 4814:5900
VKKILYIKALFLCTVIKLRRFIFSVNNMKF...DLP - length 165, strand 1, 5923:6421
LSHTVTDFTDQMAQVGLCQCVNVFLDEVTG...KAA - length 107, strand -1, 5974:6298
GCLMKKSSIVATIITILSGSANAASSQLIP...YRF - length 315, strand 1, 6654:7602
IYSTSEHTGEQVMRTLDEVIASRSPESQTR...FHV - length 111, strand -1, 7788:8124
WGKLQVIGLSMWMVLFSQRFDDWLNEQEDA...ESK - length 125, strand -1, 8087:8465
TGKQNSCQMSAIWQLRQNTATKTRQNRARI...AIK - length 100, strand 1, 8741:9044
QGSGYAFPHASILSGIAMSHFYFLVLHAVK...CSD - length 114, strand -1, 9264:9609
如果注释掉排序语句,则蛋白质序列将按之前的顺序显示,因此可以检查它是否在执行相同操作。这里我们按位置对它们进行了排序,以便更容易与 GenBank 文件中的实际注释进行比较(如第 A nice example 节中所述)。
但是,如果您只是想找到开放阅读框的位置,那么翻译每个可能的密码子(包括对反向互补链进行反向互补)就是浪费时间。您只需要搜索可能的终止密码子(及其反向互补)。使用正则表达式是这里的一个明显方法(请参阅 Python 模块 re)。这些是描述搜索字符串的极其强大的(但相当复杂)方法,它们在许多编程语言中以及 grep 等命令行工具中都得到了支持。您可以找到关于这个主题的整本书!
序列解析加上简单绘图
本节展示了更多序列解析示例,使用第 Sequence Input/Output 章中介绍的 Bio.SeqIO 模块,以及 Python 库 matplotlib 的 pylab 绘图接口(请参阅 matplotlib 网站以获取教程)。请注意,要执行这些示例,需要安装 matplotlib - 但没有它,您仍然可以尝试数据解析部分。
序列长度直方图
在许多情况下,您可能希望可视化数据集中序列长度的分布 - 例如基因组组装项目中片段的大小范围。在本例中,我们将重新使用我们的兰花 FASTA 文件 ls_orchid.fasta,它只有 94 个序列。
首先,我们将使用 Bio.SeqIO 解析 FASTA 文件并编译所有序列长度的列表。可以使用 for 循环来实现,但我发现列表推导更令人满意
>>> from Bio import SeqIO
>>> sizes = [len(rec) for rec in SeqIO.parse("ls_orchid.fasta", "fasta")]
>>> len(sizes), min(sizes), max(sizes)
(94, 572, 789)
>>> sizes
[740, 753, 748, 744, 733, 718, 730, 704, 740, 709, 700, 726, ..., 592]
现在我们有了所有基因的长度(作为整数列表),可以使用 matplotlib 直方图函数来显示它。
from Bio import SeqIO
sizes = [len(rec) for rec in SeqIO.parse("ls_orchid.fasta", "fasta")]
import pylab
pylab.hist(sizes, bins=20)
pylab.title(
"%i orchid sequences\nLengths %i to %i" % (len(sizes), min(sizes), max(sizes))
)
pylab.xlabel("Sequence length (bp)")
pylab.ylabel("Count")
pylab.show()
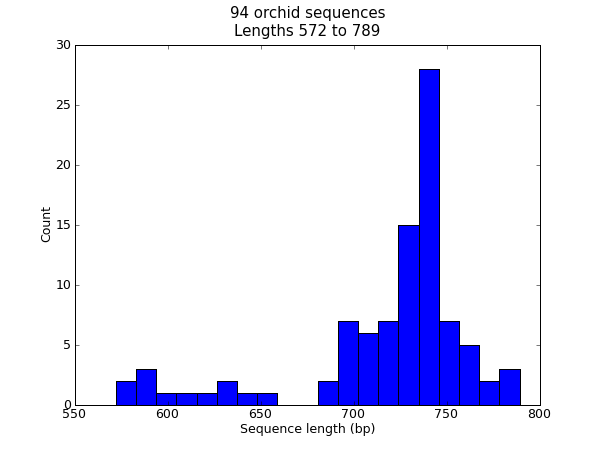
图 22 兰花序列长度直方图。
这应该会弹出一个新窗口,其中包含 图 22 中所示的图形。请注意,大多数兰花序列的长度约为 \(740\) bp,并且这里可能存在两类不同的序列,其中一类是较短的序列。
提示: 除了使用 pylab.show() 在窗口中显示绘图之外,还可以使用 pylab.savefig(...) 将图形保存到文件(例如,作为 PNG 或 PDF)。
序列 GC% 的绘图
另一个易于计算的核苷酸序列量是 GC%。例如,您可能希望查看细菌基因组中所有基因的 GC%,并调查任何可能是通过水平基因转移新获得的异常值。同样,对于此示例,我们将重新使用兰花 FASTA 文件 ls_orchid.fasta。
首先,我们将使用 Bio.SeqIO 解析 FASTA 文件并编译所有 GC 百分比的列表。同样,您也可以使用 for 循环来执行此操作,但我更喜欢这种方法
from Bio import SeqIO
from Bio.SeqUtils import gc_fraction
gc_values = sorted(
100 * gc_fraction(rec.seq) for rec in SeqIO.parse("ls_orchid.fasta", "fasta")
)
读取每个序列并计算 GC% 后,我们将它们按升序排序。现在,我们将获取此浮点数列表并使用 matplotlib 绘制它们
import pylab
pylab.plot(gc_values)
pylab.title(
"%i orchid sequences\nGC%% %0.1f to %0.1f"
% (len(gc_values), min(gc_values), max(gc_values))
)
pylab.xlabel("Genes")
pylab.ylabel("GC%")
pylab.show()
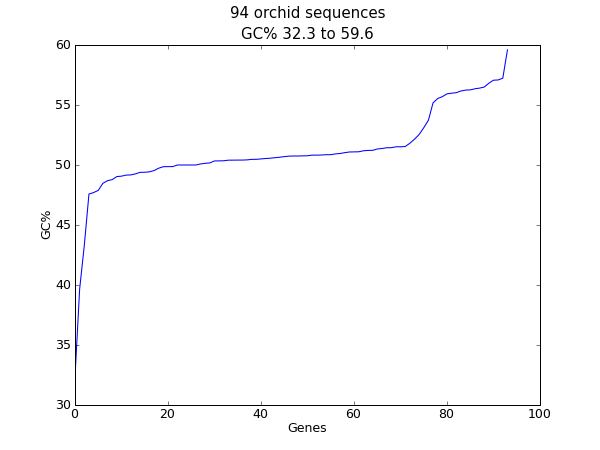
图 23 兰花序列长度直方图。
与前面的示例一样,这应该会弹出一个新窗口,其中包含 图 23 中所示的图形。如果您尝试对一个生物体的完整基因集执行此操作,您可能将获得比这更平滑的绘图。
核苷酸点图
点图是一种直观比较两个核苷酸序列相似性的方法。使用滑动窗口比较彼此的短子序列,通常使用不匹配阈值。为了简单起见,我们将只寻找完全匹配(在 图 24 中以黑色显示)。
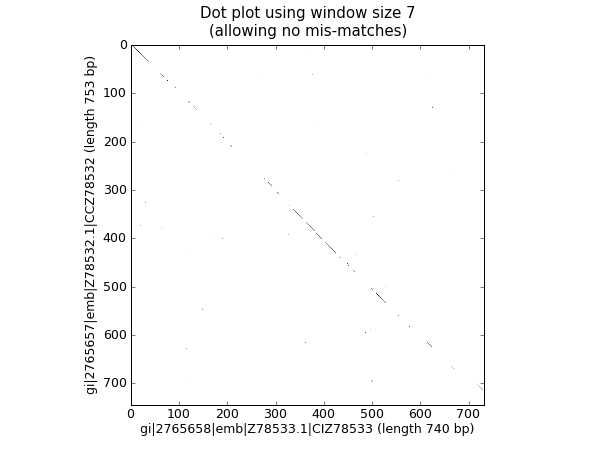
图 24 使用图像显示的两个兰花序列的核苷酸点图。
首先,我们需要两个序列。为了便于讨论,我们将从我们的兰花 FASTA 文件 ls_orchid.fasta 中获取前两个序列
from Bio import SeqIO
with open("ls_orchid.fasta") as in_handle:
record_iterator = SeqIO.parse(in_handle, "fasta")
rec_one = next(record_iterator)
rec_two = next(record_iterator)
我们将展示两种方法。首先,一个简单的朴素实现,它将所有窗口大小的子序列相互比较以编译相似性矩阵。您可以构建矩阵或数组对象,但这里我们只使用使用嵌套列表推导创建的布尔值的列表列表
window = 7
seq_one = rec_one.seq.upper()
seq_two = rec_two.seq.upper()
data = [
[
(seq_one[i : i + window] != seq_two[j : j + window])
for j in range(len(seq_one) - window)
]
for i in range(len(seq_two) - window)
]
请注意,我们尚未在此处检查反向互补匹配。现在,我们将使用 matplotlib 的 pylab.imshow() 函数来显示此数据,首先请求灰色颜色方案,因此以黑白显示
import pylab
pylab.gray()
pylab.imshow(data)
pylab.xlabel("%s (length %i bp)" % (rec_one.id, len(rec_one)))
pylab.ylabel("%s (length %i bp)" % (rec_two.id, len(rec_two)))
pylab.title("Dot plot using window size %i\n(allowing no mis-matches)" % window)
pylab.show()
这应该会弹出一个新窗口,显示 图 24 中的图形。正如您可能已经预料到的那样,这两个序列非常相似,在对角线上有一条部分窗口大小的匹配线。没有对角线外的匹配,这表明不存在倒置或其他有趣的事件。
上面的代码在小示例中效果很好,但将此代码应用于更大的序列时存在两个问题,我们将在下面讨论。首先,这种对所有与所有比较的蛮力方法非常慢。相反,我们将编译字典,将窗口大小的子序列映射到它们的位置,然后取集合交集以找到在两个序列中都找到的子序列。这会使用更多内存,但快得多。其次,pylab.imshow() 函数在可以显示的矩阵大小方面受到限制。作为替代方案,我们将使用 pylab.scatter() 函数。
我们首先创建字典,将窗口大小的子序列映射到位置
window = 7
dict_one = {}
dict_two = {}
for seq, section_dict in [
(rec_one.seq.upper(), dict_one),
(rec_two.seq.upper(), dict_two),
]:
for i in range(len(seq) - window):
section = seq[i : i + window]
try:
section_dict[section].append(i)
except KeyError:
section_dict[section] = [i]
# Now find any sub-sequences found in both sequences
matches = set(dict_one).intersection(dict_two)
print("%i unique matches" % len(matches))
为了使用 pylab.scatter(),我们需要为 \(x\) 和 \(y\) 坐标创建单独的列表
# Create lists of x and y coordinates for scatter plot
x = []
y = []
for section in matches:
for i in dict_one[section]:
for j in dict_two[section]:
x.append(i)
y.append(j)
我们现在准备绘制修订后的点图作为散点图
import pylab
pylab.cla() # clear any prior graph
pylab.gray()
pylab.scatter(x, y)
pylab.xlim(0, len(rec_one) - window)
pylab.ylim(0, len(rec_two) - window)
pylab.xlabel("%s (length %i bp)" % (rec_one.id, len(rec_one)))
pylab.ylabel("%s (length %i bp)" % (rec_two.id, len(rec_two)))
pylab.title("Dot plot using window size %i\n(allowing no mis-matches)" % window)
pylab.show()
这应该会弹出一个新窗口,显示 图 25 中的图形。
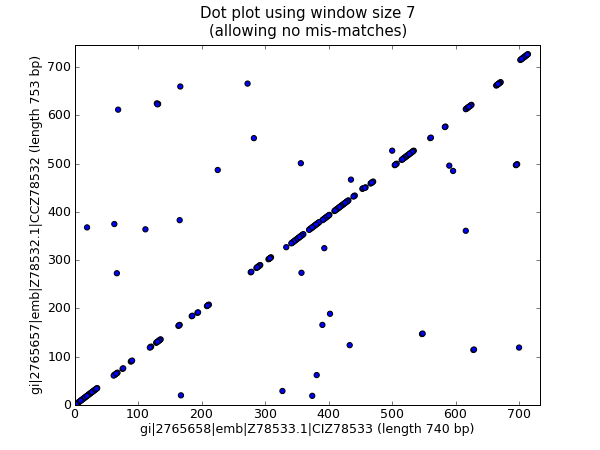
图 25 使用散点的两个兰花序列的核苷酸点图。
我个人觉得第二个绘图更容易阅读!同样请注意,我们尚未在此处检查反向互补匹配 - 您可以在此示例中扩展此功能,并将正向匹配以一种颜色绘制,反向匹配以另一种颜色绘制。
绘制测序读取数据的质量得分
如果您正在使用第二代测序数据,您可能想尝试绘制质量数据。以下是一个使用两个包含配对末端读取的 FASTQ 文件的示例,SRR001666_1.fastq 用于正向读取,SRR001666_2.fastq 用于反向读取。这些文件是从 ENA 测序读取档案 FTP 站点下载的(ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR001/SRR001666/SRR001666_1.fastq.gz 和 ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR001/SRR001666/SRR001666_2.fastq.gz),来自大肠杆菌 - 有关详细信息,请参阅 https://www.ebi.ac.uk/ena/data/view/SRR001666。
在以下代码中,pylab.subplot(...) 函数用于将正向和反向质量显示在两个子图上,并排显示。还有一些代码只绘制前五十个读取。
import pylab
from Bio import SeqIO
for subfigure in [1, 2]:
filename = "SRR001666_%i.fastq" % subfigure
pylab.subplot(1, 2, subfigure)
for i, record in enumerate(SeqIO.parse(filename, "fastq")):
if i >= 50:
break # trick!
pylab.plot(record.letter_annotations["phred_quality"])
pylab.ylim(0, 45)
pylab.ylabel("PHRED quality score")
pylab.xlabel("Position")
pylab.savefig("SRR001666.png")
print("Done")
您应该注意,我们在此处使用 Bio.SeqIO 格式名称 fastq,因为 NCBI 使用标准 Sanger FASTQ 格式(带有 PHRED 分数)保存了这些读取。但是,正如您从读取长度中可能猜到的那样,这些数据来自 Illumina Genome Analyzer,可能最初使用的是两种 Solexa/Illumina FASTQ 变体文件格式之一。
此示例使用 pylab.savefig(...) 函数而不是 pylab.show(...),但正如之前提到的,两者都有用。
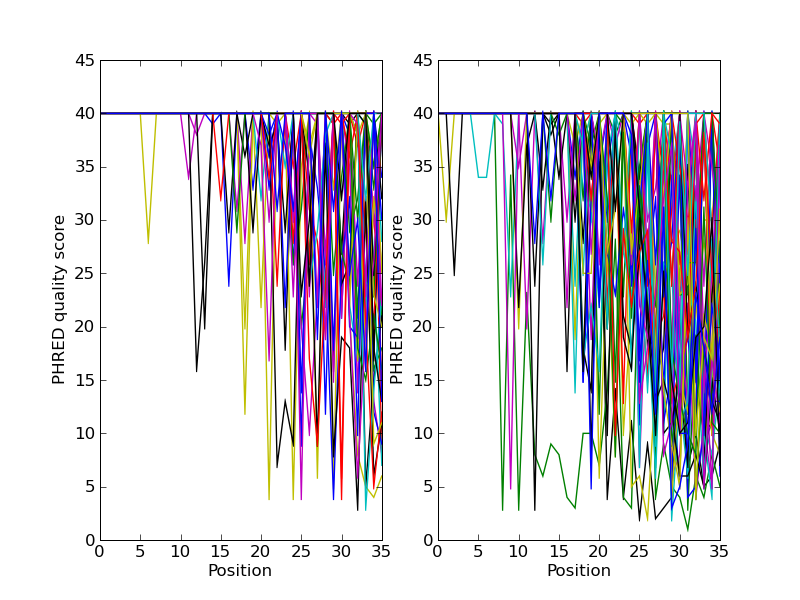
图 26 一些配对末端读取的质量图。
结果如 图 26 所示。
BioSQL - 将序列存储在关系数据库中
BioSQL 是 OBF 项目(BioPerl、BioJava 等)之间的一项共同努力,旨在支持用于存储序列数据的共享数据库模式。理论上,您可以使用 BioPerl 将 GenBank 文件加载到数据库中,然后使用 Biopython 将其从数据库中提取为具有特征的记录对象 - 并获得或多或少与您直接将 GenBank 文件加载为 SeqRecord 使用 Bio.SeqIO(第 序列输入/输出 章)时相同的内容。
Biopython 的 BioSQL 模块目前在 https://biopython.pythonlang.cn/wiki/BioSQL 中有记录,它是我们维基页面的一部分。