Bio.PDB 是一个 Biopython 模块,专注于处理生物大分子晶体结构。本文档提供了对 Bio.PDB 的相当完整的概述。
Bio.PDB 作为 Biopython 的一部分自动安装。Biopython 可从 http://www.biopython.org 获取。它运行在许多平台上(Linux/Unix、windows、Mac 等)。
Bio.PDB 用于构建 DISEMBL,这是一个预测蛋白质无序区域的 web 服务器,以及 COLUMBA,一个提供带注释蛋白质结构的网站(不再可用?)。Bio.PDB 也被用来对 PDB 中的蛋白质结构进行大规模的活性位点相似性搜索(参见 Proteins 51: 96–108, 2003),并开发了一种新的算法来识别线性二级结构元素(参见 BMC Bioinformatics 6: 202, 2005)。
从对功能和信息的请求来看,Bio.PDB 也被一些 LPC(大型制药公司 :-)使用。
是的,如果您使用了 Bio.PDB,请在出版物中引用它。参考文献是
Hamelryck, T., Manderick, B. (2003) PDB 解析器和结构类在 Python 中实现。Bioinformatics 19: 2308–2310
这篇文章可以通过 Bioinformatics 杂志网站 免费下载。欢迎您发邮件告诉我您如何使用 Bio.PDB。功能请求也欢迎。
实际上,测试得非常好。Bio.PDB 已对 PDB 中近 5500 个结构进行了广泛测试 - 所有结构似乎都解析正确。更多细节可以在 Bio.PDB Bioinformatics 文章中找到。Bio.PDB 已经/正在许多研究项目中被用作可靠工具。实际上,我几乎每天都在研究中使用 Bio.PDB,并继续努力改进它并添加新功能。
PDBParser 的性能在约 800 个结构(每个结构属于一个唯一的 SCOP 超家族)上进行了测试。这大约需要 20 分钟,平均每个结构 1.5 秒。解析大型核糖体亚基(1FKK)的结构,该结构包含约 64000 个原子,在一台 1000 MHz 的 PC 上需要 10 秒。简而言之:它对于许多应用来说已经足够快了。
Bio.PDB 可能正是您想要的,也可能不是。如果您对挖掘 PDB 标头数据感兴趣,您可能需要看看其他地方,因为对这方面支持有限。如果您需要一个功能强大、完整的结构来访问原子数据,Bio.PDB 可能适合您。
这很简单
from Bio.PDB import *
没有直接支持,主要是因为已经有很多基于 Python 或与 Python 兼容的解决方案,可以与 Bio.PDB 结合使用。我的选择是 Pymol,顺便说一下(我已经成功地将它与 Bio.PDB 结合使用,并且可能很快/有一天会在 Bio.PDB 中出现专门的 PyMol 模块)。基于 Python 或与 Python 兼容的分子图形解决方案包括
我疯了才会写另一个分子图形应用程序(已经做过,实际上 :-)
但是,您可以使用 nglview 在 Jupyter 笔记本中交互式地查看 Biopython 结构实体。
import nglview as nv
view = nv.show_biopython(structure)
view
首先,创建一个 PDBParser 对象
parser = PDBParser()
然后,以以下方式从 PDB 文件创建结构对象(本例中 PDB 文件名为 1FAT.pdb,PHA-L 是用户为结构定义的名称)
structure = parser.get_structure("PHA-L", "1FAT.pdb")
与 PDB 文件类似,首先创建一个 MMCIFParser 对象
parser = MMCIFParser()
然后使用此解析器从 mmCIF 文件创建结构对象
structure = parser.get_structure("PHA-L", "1FAT.cif")
目前还不支持,但我计划在未来支持它(这并不困难)。如果您需要此功能,请与我联系,这可能会鼓励我 :-)。
没问题。您可以创建一个 Python 字典,它将 mmCIF 文件中的所有 mmCIF 标签映射到它们的值。如果有 多个值(如标签 _atom_site.Cartn_y,它包含所有原子的 y 坐标),则标签将映射到值列表。该字典由 mmCIF 文件创建如下
from Bio.PDB.MMCIF2Dict import MMCIF2Dict
mmcif_dict = MMCIF2Dict("1FAT.cif")
示例:从 mmCIF 文件获取溶剂含量
sc = mmcif_dict["_exptl_crystal.density_percent_sol"]
示例:获取所有原子的 y 坐标列表
y_list = mmcif_dict["_atom_site.Cartn_y"]
感谢 Christian Rother,您可以访问 PDB 标头中的一些信息。但是请注意,许多 PDB 文件包含标头,其中包含不完整或错误的信息。许多错误已在等效的 mmCIF 文件中得到修复。**因此,如果您对标头信息感兴趣,建议您使用上面描述的 MMCIF2Dict 工具从 mmCIF 文件中提取信息,而不是解析 PDB 标头。**
现在已经澄清了这一点,让我们回到解析 PDB 标头。结构对象有一个名为 header 的属性,它是一个 python 字典,将标头记录映射到它们的值。
示例
resolution = structure.header["resolution"]
keywords = structure.header["keywords"]
可用的键是 name、head、deposition_date、release_date、structure_method、resolution、structure_reference(映射到引用列表)、journal_reference、author 和 compound(映射到一个包含有关结晶化合物的各种信息的字典)。
该字典也可以在不创建 Structure 对象的情况下创建,即直接从 PDB 文件创建
handle = open(filename, "r")
header_dict = parse_pdb_header(handle)
handle.close()
当然可以。许多 PDB 解析器假设只有一个模型,这使得它们对于 NMR 结构几乎毫无用处。Structure 对象的设计使得它易于处理具有多个模型的 PDB 文件(参见 结构对象 部分)。
这可以通过使用 PDBList 对象和 retrieve_pdb_file 方法来完成。此方法的参数是结构的 PDB 标识符。
pdbl = PDBList()
pdbl.retrieve_pdb_file("1FAT")
PDBList 类也可以用作命令行工具
python PDBList.py 1fat
下载的文件将被称为 pdb1fat.ent 并存储在当前工作目录中。请注意,retrieve_pdb_file 方法还有一个可选参数 pdir,它指定存储下载的 PDB 文件的特定目录。
retrieve_pdb_file 方法还有一些选项用于指定用于下载的压缩格式以及用于本地解压缩的程序(默认情况下为 .Z 格式和 gunzip)。此外,在创建 PDBList 对象时可以指定 PDB ftp 站点。默认情况下,使用 全球蛋白质数据库 的 ftp 服务器。有关更多详细信息,请参见 API 文档。再次感谢 Kristian Rother 为此模块捐赠了代码。
以下命令将所有 PDB 文件存储在 /data/pdb 目录中
python PDBList.py all /data/pdb
python PDBList.py all /data/pdb -d
此方法的 API 方法称为 download_entire_pdb。添加 -d 选项将把所有文件存储在同一个目录中。否则,它们将根据其 PDB ID 排序到 PDB 样式的子目录中。根据流量,完整下载将花费 2-4 天。
这也可以使用 PDBList 对象来完成。只需创建一个 PDBList 对象(指定 PDB 本地副本所在的目录)并调用 update_pdb 方法
pl = PDBList(pdb="/data/pdb")
pl.update_pdb()
当然,您可以将此操作设置为每周的 cron 作业,以便自动更新本地副本。也可以指定 PDB ftp 站点(参见 API 文档)。
PDBList 有一些其他方法可能有用。可以使用 get_all_obsolete 方法获取所有已废弃 PDB 条目的列表。可以使用 changed_this_week 方法获取本周添加、修改或废弃的条目。有关 PDBList 的更多信息,请参见 API 文档。
众所周知,许多 PDB 文件包含语义错误(我指的不是结构本身,而是它们在 PDB 文件中的表示)。Bio.PDB 尝试通过两种方式来处理这个问题。PDBParser 对象可以以两种方式运行:一种是严格的方式,另一种是宽松的方式(现在是默认方式)。严格的方式曾经是默认方式,但人们似乎认为 Bio.PDB '崩溃' 了,是因为存在一个 bug(哈哈!),所以我把它改了。如果您遇到真正的 bug,请立即告诉我!
示例
# Permissive parser
parser = PDBParser(PERMISSIVE=1)
parser = PDBParser() # The same (default)
# Strict parser
strict_parser = PDBParser(PERMISSIVE=0)
在允许状态 (DEFAULT) 下,明显包含错误的 PDB 文件会被“修正”(即,一些残基或原子会被省略)。这些错误包括:
这些错误表明 PDB 文件中存在实际问题(有关详细信息,请参阅生物信息学文章)。在限制状态下,包含错误的 PDB 文件会导致异常发生。这有助于查找 PDB 文件中的错误。
但是,某些错误会自动更正。通常,每个无序原子都应该具有一个非空白的 altloc 标识符。但是,许多结构不遵循此约定,并且对于同一原子的两个无序位置,具有空白和非空白标识符。系统会自动将此解释为正确的方式。
有时,结构包含属于链 A 的残基列表,然后是属于链 B 的残基,然后又是属于链 A 的残基,即链是“断裂”的。这也被正确解释。
为此,请使用 PDBIO 类。当然,也可以轻松地写入结构的特定部分。
示例:保存结构
io = PDBIO()
io.set_structure(s)
io.save("out.pdb")
如果要写入结构的一部分,请使用 Select 类(也在 PDBIO 中)。Select 有四种方法
accept_model(model)
accept_chain(chain)
accept_residue(residue)
accept_atom(atom)
默认情况下,每个方法都返回 1(表示模型/链/残基/原子包含在输出中)。通过子类化 Select 并在合适的情况下返回 0,可以将模型、链等从输出中排除。可能很麻烦,但非常强大。以下代码只写入甘氨酸残基
class GlySelect(Select):
def accept_residue(self, residue):
if residue.get_name() == "GLY":
return 1
else:
return 0
io = PDBIO()
io.set_structure(s)
io.save("gly_only.pdb", GlySelect())
如果这一切对你来说太复杂了,Dice 模块包含一个方便的 extract 函数,可以写入链中从起始残基到结束残基的所有残基。
不,我目前也没有计划很快(甚至永远)添加该功能(我根本不需要它,而且工作量很大,而且没有人要求过它)。想要添加此功能的人可以联系我。
The Structure object follows the so-called SMCRA (Structure/Model/Chain/Residue/Atom) architecture
这是许多结构生物学家/生物信息学家看待结构的方式,它提供了一种简单但高效的方式来处理结构。在需要时,会添加额外的内容。下图显示了 Structure 对象的 UML 图(目前忽略 Disordered 类)。
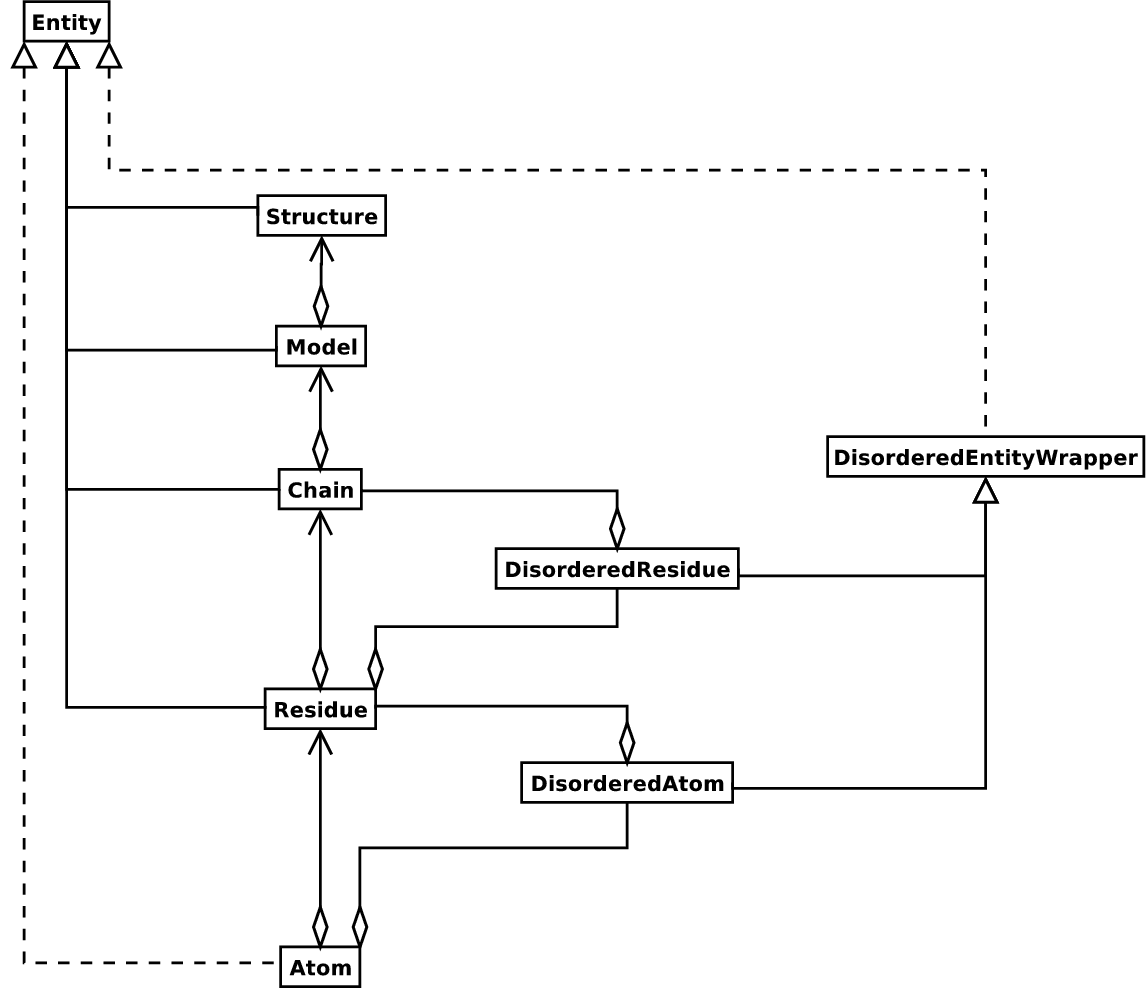
结构对象的 SMCRA 架构图。
带有菱形的实线表示聚合,带有箭头的实线表示引用,带有三角形的实线表示继承,带有三角形的虚线表示接口实现。
以下代码遍历结构的所有原子
p = PDBParser()
structure = p.get_structure("X", "pdb1fat.ent")
for model in structure:
for chain in model:
for residue in chain:
for atom in residue:
print(atom)
还有一些快捷方式
# Iterate over all atoms in a structure
for atom in structure.get_atoms():
print(atom)
# Iterate over all residues in a model
for residue in model.get_residues():
print(residue)
结构、模型、链、残基和原子在 Biopython 中被称为 **实体**。始终可以从子 **实体** 获取父 **实体**,例如:
residue = atom.get_parent()
chain = residue.get_parent()
还可以使用 has_id 方法测试 **实体** 是否具有某个特定子项。
你可以做以下事情
atoms = structure.get_atoms()
residues = structure.get_residues()
atoms = chain.get_atoms()
你也可以使用 Selection.unfold_entities 函数
# Get all residues from a structure
res_list = Selection.unfold_entities(structure, "R")
# Get all atoms from a chain
atom_list = Selection.unfold_entities(chain, "A")
显然,A=原子,R=残基,C=链,M=模型,S=结构。你可以使用它在层次结构中向上移动,例如,从原子列表获取(唯一)Residue 或 Chain 父项
residue_list = Selection.unfold_entities(atom_list, "R")
chain_list = Selection.unfold_entities(atom_list, "C")
有关更多信息,请参见 API 文档。
Structure 中提取特定的 Atom/Residue/Chain/Model?很简单。以下是一些示例
model = structure[0]
chain = model["A"]
residue = chain[100]
atom = residue["CA"]
请注意,可以使用快捷方式
atom = structure[0]["A"][100]["CA"]
模型 ID 是一个整数,表示模型在 PDB/mmCIF 文件中的排名。模型 ID 从 0 开始。晶体结构通常只有一个模型(ID 为 0),而 NMR 文件通常有多个模型。
链 ID 在 PDB/mmCIF 文件中指定,是一个单字符(通常是一个字母)。
由于 PDB 格式笨拙,这有点复杂。残基 ID 是一个包含三个元素的元组
'H_' 加上异构体残基的名称(例如,葡萄糖分子的 'H_GLC'),或 'W'(水分子)。'A'。插入代码有时用于保留某些理想的残基编号方案。Ser 80 插入突变体(例如,插入在 Thr 80 和 Asn 81 残基之间)可能具有以下序列标识符和插入代码:Thr 80 A、Ser 80 B、Asn 81。通过这种方式,残基编号方案与野生型结构的编号方案保持一致。因此,上述葡萄糖残基的 ID 为 ('H_GLC', 100, 'A')。如果异构体标志和插入代码为空白,则可以使用序列标识符本身
# Full id
residue = chain[(" ", 100, " ")]
# Shortcut id
residue = chain[100]
使用异构体标志的原因是,许多 PDB 文件对氨基酸和异构体残基或水使用相同的序列标识符,如果未使用异构体标志,这会导致明显的问题。
原子 ID 就是原子名称(例如,'CA')。实际上,原子名称是通过从 PDB 文件中的原子名称中删除所有空格创建的。
但是,在 PDB 文件中,空格可以是原子名称的一部分。通常,钙原子被称为 'CA..',以便将其与 Cα 原子(称为 '.CA.')区分开来。在删除空格会导致问题(即,同一残基中存在两个名为 'CA' 的原子)的情况下,会保留空格。
这是 Bio.PDB 的优势之一。它可以同时处理无序原子和点突变(即,同一位置的 Gly 和 Ala 残基)。
应从两个方面处理无序:原子和残基的视角。总的来说,我尝试封装了无序产生的所有复杂性。如果你只想遍历所有 Cα 原子,你不会关心某些残基是否具有无序侧链。另一方面,也应该能够在数据结构中完全表示无序。因此,无序原子或残基存储在特殊对象中,这些对象的行为就像没有无序一样。这是通过仅表示无序原子或残基的子集来实现的。用户可以指定选择哪个子集(例如,使用 Ser 残基的两个无序 OG 侧链原子位置中的哪一个)。
无序原子位置由普通的 Atom 对象表示,但所有代表相同物理原子的 Atom 对象都存储在 DisorderedAtom 对象中(参见部分 结构对象)。DisorderedAtom 对象中的每个 Atom 对象都可以使用其 altloc 说明符进行唯一索引。The DisorderedAtom object forwards all uncaught method calls to the selected Atom object, by default the one that represents the atom with the highest occupancy. 用户当然可以更改选定的 Atom 对象,利用其 altloc 说明符。通过这种方式,原子无序可以得到正确表示,而不会增加太多复杂性。换句话说,如果你对原子无序不感兴趣,你就不会受到它的困扰。
每个无序原子都有一个特征性的 altloc 标识符。你可以指定 DisorderedAtom 对象应该像与特定 altloc 标识符关联的 Atom 对象一样工作
atom.disordered_select("A") # select altloc A atom
atom.disordered_select("B") # select altloc B atom
当无序是由 **点突变** 引起的时,会出现特殊情况,即当多肽的两个或多个点突变体存在于晶体中时。在 PDB 结构 1EN2 中可以找到此示例。
由于这些残基属于不同的残基类型(例如,假设是 Ser 60 和 Cys 60),因此它们不应像常见情况下那样存储在一个 Residue 对象中。在这种情况下,每个残基由一个 Residue 对象表示,并且这两个 Residue 对象存储在一个 DisorderedResidue 对象中(参见部分 结构对象)。
The DisorderedResidue object forwards all uncaught methods to the selected Residue object (by default the last Residue object added), and thus behaves like an ordinary residue. DisorderedResidue 对象中的每个 Residue 对象都可以通过其残基名称进行唯一标识。在上面的示例中,Ser 60 残基在 DisorderedResidue 对象中的 ID 为‘SER’,而 Cys 60 残基的 ID 为‘CYS’。用户可以通过此 ID 选择 DisorderedResidue 对象中的活动 Residue 对象。
示例:假设一条链在位置 10 处有一个点突变,由一个 Ser 残基和一个 Cys 残基组成。确保该链的残基 10 作为 Cys 残基工作。
residue = chain[10]
residue.disordered_select("CYS")
此外,您可以使用 (Disordered)Residue 对象的 get_unpacked_list 方法获取所有 Atom 对象的列表(即,所有 DisorderedAtom 对象都“解包”到它们各自的 Atom 对象中)。
可以,有点,但我正在等待一个针对此功能的请求才能完成它 :-)。
参见 ‘残基 ID 是什么?’。
当然可以!Bio.PDB 支持各向同性和各向异性 B 因子,并且还处理各向异性 B 因子的标准偏差(如果存在)(参见部分 分析)。
是的,支持。参见部分 分析。
当然,当然。每个人都在不断地提出(主要是半成品或部分实现的)数据结构,这些结构可以处理所有可能的情况,并且可以在所有可以想到的(和不可想象的)方式中扩展。然而,平淡的事实是,99.9% 使用(我指的是真正使用!)晶体结构的人,都是从模型、链、残基和原子的角度思考问题。 Bio.PDB 的理念是提供一个合理快速的、简洁的、简单的、但完整的用于访问结构数据的结构。实践是检验真理的唯一标准。
此外,在 Structure 类之上构建更专业化的数据结构非常容易(例如,有一个 Polypeptide 类)。另一方面,Structure 对象是使用解析器/使用者方法构建的(分别称为 PDBParser/MMCIFParser 和 StructureBuilder)。通过实现一个专门的 StructureBuilder 类,可以轻松地重用 PDB/mmCIF 解析器。当然,通过编写新的解析器,也可以轻松地添加对新文件格式的支持。
Atom 对象中提取信息?使用以下方法
a.get_name() # atom name (spaces stripped, e.g. 'CA')
a.get_id() # id (equals atom name)
a.get_coord() # atomic coordinates
a.get_vector() # atomic coordinates as Vector object
a.get_bfactor() # isotropic B factor
a.get_occupancy() # occupancy
a.get_altloc() # alternative location specifier
a.get_sigatm() # std. dev. of atomic parameters
a.get_siguij() # std. dev. of anisotropic B factor
a.get_anisou() # anisotropic B factor
a.get_fullname() # atom name (with spaces, e.g. '.CA.')
Residue 对象中提取信息?使用以下方法
r.get_resname() # return the residue name (eg. 'GLY')
r.is_disordered() # 1 if the residue has disordered atoms
r.get_segid() # return the SEGID
r.has_id(name) # test if a residue has a certain atom
很简单:原子的减法运算符已被重载,以返回两个原子之间的距离。
示例
# Get some atoms
ca1 = residue1["CA"]
ca2 = residue2["CA"]
# Simply subtract the atoms to get their distance
distance = ca1 - ca2
这可以通过原子坐标的向量表示以及 Vector 模块的 calc_angle 函数轻松完成。
vector1 = atom1.get_vector()
vector2 = atom2.get_vector()
vector3 = atom3.get_vector()
angle = calc_angle(vector1, vector2, vector3)
同样,这可以通过原子坐标的向量表示轻松完成,这次使用 Vector 模块的 calc_dihedral 函数。
vector1 = atom1.get_vector()
vector2 = atom2.get_vector()
vector3 = atom3.get_vector()
vector4 = atom4.get_vector()
angle = calc_dihedral(vector1, vector2, vector3, vector4)
使用 NeighborSearch。这在幕后使用 C 语言编写的 KD 树数据结构,因此速度非常快(参见 Bio.KDTree)。
Structure 对象中提取多肽?使用 PolypeptideBuilder。您可以使用生成的 Polypeptide 对象获取作为 Seq 对象的序列,或者获取 Cα 原子的列表。多肽可以使用 C-N 或 Cα-Cα 距离准则构建。
示例
# Using C-N
ppb = PPBuilder()
for pp in ppb.build_peptides(structure):
print(pp.get_sequence())
# Using CA-CA
ppb = CaPPBuilder()
for pp in ppb.build_peptides(structure):
print(pp.get_sequence())
请注意,在上述情况下,PolypeptideBuilder 仅考虑结构的模型 0。但是,可以使用 PolypeptideBuilder 从 Model 和 Chain 对象构建 Polypeptide 对象。
首先要做的是从结构中提取所有多肽(参见上一条条目)。然后可以轻松地从 Polypeptide 对象获得每个多肽的序列。序列表示为 Biopython Seq 对象。
示例
>>> seq = polypeptide.get_sequence()
>>> print(seq)
Seq('SNVVE...')
要实现此功能,您需要安装 DSSP(并获得其许可 - 学术用途免费)。然后使用 DSSP 类,它将 Residue 对象映射到它们的二级结构(以及可及表面积)。DSSP 代码列在下面的表格中。请注意,DSSP(程序,因此是类)无法处理多个模型!
| DSSP 代码 | 二级结构 |
|---|---|
| H | α-螺旋 |
| B | 孤立的 β-桥残基 |
| E | 链 |
| G | 3-10 螺旋 |
| I | Π-螺旋 |
| T | 转角 |
| S | 弯曲 |
| - | 其他 |
使用 DSSP 类(参见上一条条目)。但是,另请参见下一条条目。
残基深度是残基原子到溶剂可及表面的平均距离。这是一个相当新的、非常强大的溶剂可及性参数化方法。要实现此功能,您需要安装 Michel Sanner 的 MSMS 程序。然后使用 ResidueDepth 类。此类充当字典,它将 Residue 对象映射到相应的(残基深度、Cα 深度)元组。Cα 深度是残基的 Cα 原子到溶剂可及表面的距离。
示例
model = structure[0]
rd = ResidueDepth(model, pdb_file)
residue_depth, ca_depth = rd[some_residue]
您还可以通过 get_surface 函数访问分子表面本身,形式为包含表面点的 NumPy 数组。
半球暴露 (HSE) 是一种新的 2D 溶剂暴露度量。基本上,它计算残基侧链方向和相反方向(在 13 Å 半径内)周围的 Cα 原子数量。尽管它很简单,但它优于许多其他溶剂暴露度量。描述这种新型 2D 度量的文章已提交发表。
HSE 有两种形式:HSEα 和 HSEβ。前者仅使用 Cα 原子位置,而后者使用 Cα 和 Cβ 原子位置。HSE 度量由 HSExposure 类计算,该类还可以计算接触数。后者类具有返回字典的方法,这些字典将 Residue 对象映射到其相应的 HSEα、HSEβ 和接触数的值。
示例
model = structure[0]
hse = HSExposure()
# Calculate HSEalpha
exp_ca = hse.calc_hs_exposure(model, option="CA3")
# Calculate HSEbeta
exp_cb = hse.calc_hs_exposure(model, option="CB")
# Calculate classical coordination number
exp_fs = hse.calc_fs_exposure(model)
# Print HSEalpha for a residue
print(exp_ca[some_residue])
首先,创建一个 FASTA 格式的对齐文件,然后使用 StructureAlignment 类。此类还可用于对齐两个以上的结构。
使用 is_aa(residue)。
Atom 对象使用 get_vector 方法返回坐标的 Vector 对象表示形式。 Vector 实现完整的 3D 向量运算集、矩阵乘法(左乘和右乘)以及一些高级的旋转相关运算。另请参见下一个问题。
好吧,我承认,这个示例仅仅是为了展示 Bio.PDB 的 Vector 模块的可能性(尽管此代码实际上在 HSExposure 模块中使用,该模块包含一种新的参数化残基暴露方法 - 正在发表)。假设您想找到 Gly 残基的 Cβ 原子的位置,如果它有的话。您将如何做到这一点?嗯,将 Gly 残基的 N 原子沿 Cα-C 键旋转 -120 度,大致可以将其放在虚拟 Cβ 原子的位置。以下是使用 Vector 模块的 rotaxis 方法(可用于构建围绕某个轴的旋转)来执行此操作的方法。
# get atom coordinates as vectors
n = residue["N"].get_vector()
c = residue["C"].get_vector()
ca = residue["CA"].get_vector()
# center at origin
n = n - ca
c = c - ca
# find rotation matrix that rotates n -120 degrees along the ca-c vector
rot = rotaxis(-pi * 120.0 / 180.0, c)
# apply rotation to ca-n vector
cb_at_origin = n.left_multiply(rot)
# put on top of ca atom
cb = cb_at_origin + ca
此示例表明,可以对原子数据执行一些相当非平凡的向量运算,这非常有用。除了所有常用的向量运算(叉积(使用 **)和点积(使用 *)运算、角度、范数等)以及上面提到的 rotaxis 函数外,Vector 模块还具有用于将一个向量旋转 (rotmat) 或反射 (refmat) 到另一个向量之上的方法。
令人惊讶的是,这是使用 Superimposer 对象完成的。该对象计算旋转和平移矩阵,该矩阵以使 RMSD 最小化的方式将两个原子列表彼此旋转。当然,两个列表需要包含相同数量的原子。 Superimposer 对象也可以将旋转/平移应用于原子列表。旋转和平移存储为 Superimposer 对象的 rotran 属性中的元组(请注意,旋转是右乘的!)。RMSD 存储在 rmsd 属性中。
Superimposer 使用的算法来自 矩阵计算,第二版。Golub, G. & Van Loan (1989) 并利用奇异值分解(这在通用的 Bio.SVDSuperimposer 模块中实现)。
示例
sup = Superimposer()
# Specify the atom lists
# 'fixed' and 'moving' are lists of Atom objects
# The moving atoms will be put on the fixed atoms
sup.set_atoms(fixed, moving)
# Print rotation/translation/rmsd
print(sup.rotran)
print(sup.rms)
# Apply rotation/translation to the moving atoms
sup.apply(moving)
很简单。使用活性位点原子计算旋转/平移矩阵(参见上文),并将这些矩阵应用于整个分子。
可以,使用 Atom 对象的 transform 方法,或者直接使用 set_coord 方法。
参见主要的 Biopython 教程。
目前还没有文档。
哇!已经很晚了,我累了,一杯上等的佩德罗希梅内斯雪利酒正等着我。请给我发邮件,我会在早上(如果运气好的话)回复您。
Bio.PDB 的主要作者/维护者是
Thomas Hamelryck 生物信息学中心 分子生物学研究所 哥本哈根大学 Universitetsparken 15,Bygning 10 DK-2100 København Ø 丹麦
Kristian Rother 捐赠了与 PDB 数据库交互以及解析 PDB 标头的代码。Indraneel Majumdar 提交了一些错误报告,并协助编写了 Polypeptide 模块。非常感谢 Brad Chapman、Jeffrey Chang、Andrew Dalke 和 Iddo Friedberg 的建议、评论、帮助和/或批评 :-)。
当然可以!如果您有有用的贡献,请发送电子邮件至 Thomas Hamelryck 或 Biopython 开发者!永恒的荣誉在等着您!